Вирус SARS-CoV-2 является седьмым из числа известных коронавирусов, которые способны заражать человека (коронавирусы человека). Четыре из этих вирусов - 229E, NL63, HKU1 и OC43 - являются эндемичными сезонными вирусами, которые, как правило, вызывают респираторные заболевания легкого течения. В 2002 г. и 2012 г. появились вирусы SARS-CoV и MERS-CoV, которые способны вызывать тяжелый острый респираторный синдром (ТОРС) и отнесены ко II группе патогенности. С декабря 2019 г. распространение получил новый короновирус SARS-CoV-2, а 11 марта 2020 г. ВОЗ объявила о начале пандемии COVID-19. SARS-CoV-2 включен в перечень заболеваний, представляющих опасность для окружающих (постановление Правительства Российской Федерации от 31 января 2020 г. № 66). В настоящее время диагностика острой инфекции, вызванной вирусом SARS-CoV-2, основана на методе полимеразной цепной реакции с обратной транскрипцией. В качестве мишеней обычно используются области генов вируса N, E, S и RdRP.
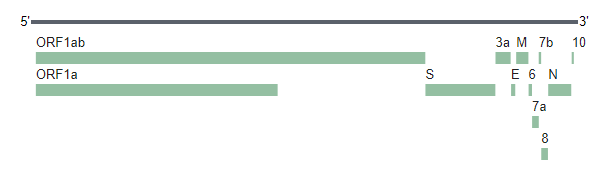
SARS-CoV-2 имеет одноцепочечную положительную нить РНК длиной примерно 30 000 п.о. Геном вируса кодирует 27 белков, включая РНК-зависимую РНК полимеразу (RdRP) и 4 структурных белка: S - белок шипа, E - белок оболочки, M - белок мембраны и N - нуклеокапсидный белок (Рис. 1). В начале января 2020 г была получена первая последовательность генома SARS-CoV2 (MN908947). По мере распространения вируса, начинают создаваться и пополняться базы данных генетических последовательностей новых штаммов (NCBI, GISAID). Эти базы данных рекомендуется использовать для мониторинга мутаций, потенциально влияющих на эффективность теста.
ПЦР-системы
Первая тест-системой для определения вируса была разработана в Институте Вирусологии в Берлине еще до получения образцов вируса [1]. Corman et al. смоделировал геном SARS-CoV-2 на основе генома SARS-CoV-1 и экспериментально подтвердили работоспособность системы. Для рутинного процесса был рекомендован анализ гена E в качестве инструмента скрининга первой линии с последующим подтверждающим тестированием с помощью анализа гена RdRp. Использование двухканальной системы детектирования при определении гена RdRp позволяет отличить SARS-Cov-2 (оба канала дают положительный результат) от SARS-CoV-1. В качестве альтернативы лаборатории могут выбрать проведение анализа RdRp со специфичным для SARS-CoV-2 зондом. В данной работе выбрана последовательность праймеров для определения гена E, которая почти полностью комплементарна последовательности SARS-CoV-1 за исключением первого нуклеотида в прямом праймере, который слабо влияет на эффективность амплификации. Таким образом, диагностическая стратегия в этом тесте основана на том, что атипичная пневмония была ликвидирована у людей (последний зарегистрированный человек с ОРВИ был выявлен в 2004 году) и результаты положительные по гену E могут считаться положительными на наличие SARS-CoV-2.
К настоящему времени доступно большое количество ПЦР-тестов как для использования внутри организаций, так и в коммерческой продаже. Перечень зарегистрированных в РФ диагностических наборов реагентов для выявления РНК SARS-CoV-2 представлен в Государственном реестре медицинских изделий. С Оптимальным считается использование системы, определяющей не менее двух независимых мишеней в геноме вируса [2]. Такой подход снижает вероятность получения ложноотрицательных результатов вследствии появления мутаций, которые могут затрагивать места гибридизации праймеров. Не все мутации в областях связывания праймера/зонда приводят к существенному изменению эффективности теста. Для подтверждения чувствительности используемой тест-системы необходима не только теоретическая, но и экспериментальная оценка эффективности гибридизаци. При использовании коммерческих тестов крайне важно принимать во внимание возможность получения неудовлетворительных результатов.
Тереоретическая оценка специфичности гибридизации праймеров является важным этапом в разработке тест-систем. В работе [3] изложен комплексный подход к биоинформатической оценке ПЦР-диагностических анализов на SARS-CoV-2. Применение этой стратегии к 27 ранее разработанных тест-систем с использованием 17 027 вирусных последовательностей показали мутации/несоответствия в областях связывания праймера/зонда в семи наборах праймеров и зондах. Независимо от этой работы, T. Pillonel также обнаружил в тест-системе на ген RdRp, разработанной Corman et al., несоответствие которое затрагивает 1 623 последовательности, опубликованных в NCBI [4]. Замена S(G/C) на R (A/G) в обратном праймере CARATGTTAAA(S→R)ACACTATTAGCATA позводяет добиться 100% чувствительности. Эти работы и примеры показывают важность постоянного мониторинга мутаций в местах связывания праймеров.

В рабое Li at al. [5] проведен обзор основных этапов в разработке SARS-CoV-2 тест-систем: определение целевой последовательности, принципов подбора последовательности праймеров и зондов, подбор вырожденных последовательностей для учета вариаций в геноме вируса. Предполагается, что более консервативный ген Е является мишенью для пан-коронавирусного анализа, в то время как гены N и RdRp используются в качестве мишеней для подтверждающих анализов. В работе показана схема разработки тест-систем (Рис. 2), включающая как теоретические, так и практические этапы оценки диагностических характеристик систем.
Литература
- V. Corman, O. Landt, M. Kaiser, R. Molenkamp, A. Meijer, D. Chu, T. Bleicker, S. Brünink, J. Schneider, M. Schmidt, D. Mulders, B. Haagmans, B. van der Veer, S. van den Brink, L. Wijsman, G. Goderski, J. Romette, J. Ellis, M. Zambon, M. Peiris, H. Goossens, C. Reusken, M. Koopmans, C. Drosten / Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR // Euro Surveillance, 2020, 25(3)
- Диагностическое тестирование для определения вируса SARS-CoV-2: Временные рекомендации, 11 сентября 2020 г.
- K. Khan, P. Cheung / Presence of mismatches between diagnostic PCR assays and coronavirus SARS-CoV-2 genome // Royal society open science, 2020, 7(6)
- P. Trestan, S. Valentin, J. Katia, G. Gilbert, B. Claire / Letter to the editor: SARS-CoV-2 detection by real-time RT-PCR // Euro Surveillance, 2020, 25(21)
- D. Li, J. Zhang, J. Li / Primer design for quantitative real-time PCR for the emerging Coronavirus SARS-CoV-2 // Theranostics, 2020, 10(16), 7150-7162
