Развитие полимеразной цепной реакции очень похоже на развитие интернета. Эти два открытия произошли примерно в одно время и уже успели несколько раз выйти за границы текущих представлений и возможностей. Их роднит и то, что те задачи, которые они решили в момент открытия, пренебрежимо малы по сравнению с теми возможностями, которые они дали для развития технологии, науки и общества. Ключевыми технологиями, которые предопределили распространение ПЦР, являются отрытие термостабильной ДНК-полимеразы, ПЦР в режиме реального времени, мультиплексная и цифровая ПЦР, ПЦР с обратной транскрипцией.
Предпосылки открытия
Первоначальная концепция ПЦР, как и многих других открытий, была комбинацией нескольких существующих техник и принципов:
- В 1957г. А.Корнберг описал фермент, выделенный из кишечной палочки, который синтезирует нить ДНК из отдельных дезоксирибонуклеотидтрифосфатов [1];
- В 1970г. К.Клеппе применил синтетические олигонуклеотиды в качестве затравки для синтеза комплементарной последовательности ДНК [2];
- В 1976г. Дж.Трела выделил термостабильную ДНК-полимеразу из термофильных бактерий Thermus Fquaticus, для которой оптимальной температурой синтеза является 80°С [3].
Эти работы позже стали основанием для патентных споров.
Открытие принципов полимеразной цепной реакции

В начале 80-х в компании Cetus в Калифорнии проводились эксперименты, кульминацией которых весной 1983 явились сформулированные Кэрри Муллисом основные принципы полимеразной цепной реакции [4]. За это открытие в 1993г. Муллис получил нобелевскую премию по химии.
Изобретением Мюллиса было использование сразу двух праймеров для того, чтобы специфично амплифицировать определенный участок ДНК. В этом случае праймеры комплементарны противоположным цепям ДНК и ограничивают амплифицируемый участок. Один из праймеров является затравкой для синтеза комплементарной нити ДНК, которая на следующем этапе будет субстратом для синтеза комплементарной нити ДНК с участием другого праймера [5]. Предложенная система подразумевает использование продуктов одного этапа синтеза комплементарной последовательности как субстрат на следующих этапах, формируя цепную реакцию.
В 1989 г. компания DuPont подала иск к компании Cetus о неправомерном получении патента, в котором утверждала, что ПЦР была уже была описана в работах Клеппе [3],Олсон [6] и Хорана [7]. Спустя 2 года суд подтвердил патент Cetus, который в этом же году был продан Hoffman-La Roche за $300 млн [8].
Термостабильная ДНК-полимераза
Основным недостатком фрагмента Кленова, который использовался Муллисом в первых экспериментах по ПЦР, является его термолабильность [9]. Это приводило к необходимости добавлять свежую партию фермента после каждого цикла денатурации. Кроме явного недостатка - высокой трудоемкости анализа, необходимость регулярного открытия пробирки приводит к риску контаминации реактивов и лаборатории. Использование термостабильной ДНК-полимеразы позволило решить сразу две проблемы [10]. Температурная стабильность этого фермента позволила проводить ПЦР в закрытой пробирке, а высокая оптимальная температура синтеза позволила значительно увеличить селективность анализа.
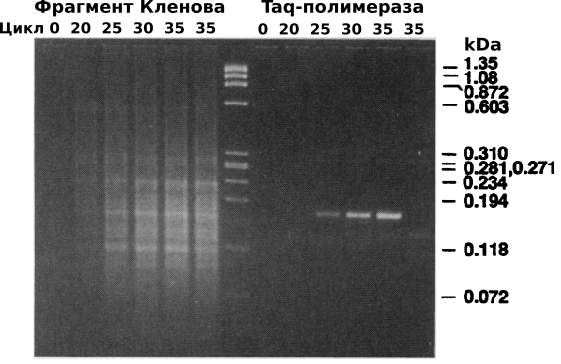
Специфичность амплификации определенного участка ДНК, задается нуклеотидной последовательностью праймеров. Чем длинее праймеры, тем они более специфичны к ДНК. Но при увеличении длины уменьшается значимость небольших несоответствий в последовательности праймера и комплементарного участка ДНК. Соответственно, на практике при использовании фрагмента Кленова и низких температур на стадии гибридизации праймеров и элонгации, увеличивается доля неспецифичесных продуктов, которые являются результатом гибридизации праймеров на не полностью соответствующий участок ДНК. Специфичность гибридизации можно повысить путем повышения температуры раствора. Замена фрагмента Кленова на Taq-полимеразу позволило реализовать эту идею и значительно увеличить специфичность амплификации.
Снижение трудоемкости и увеличение селективности анализа значительно подогрело интерес к ПЦР, но для широкого распространения ПЦР в практические сферы деятельности недоставало еще одного фактора - удобного метода детектирования продуктов реакции.
ПЦР в режиме реального времени
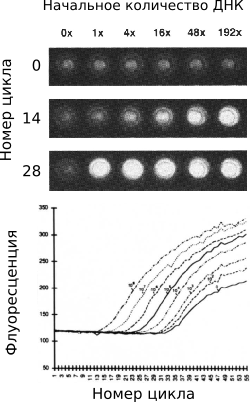
Открытие термостабильной ДНК-полимеразы позволило провести ПЦР в закрытой пробирки, но для детектирования продуктов ПЦР пробирку все-таки приходилось открывать. Это значительно ограничивало практические диагностические возможности метода, так как значительно повышало риск контаминации лаборатории. Поэтому Том Вайт, вице-президент исследовательского отдела Cetus, озвучил цель детектирования ампликонов в закрытой пробике [11].
Рус Хигучи, за которым признано открытие "кинетической" ПЦР, в это время работал в исследовательской группе над идеей ДНК-меток для прослеживания ценных или важных предметов, например, денег, взрывчатых или лекарственных веществ. Одним из побочных продуктов их экспериментов было наблюдение, что этидиум бромид, добавленный в пробирку перед проведением ПЦР, не вызывает ингибирования реакции, а количество ампликонов достаточно для детектирования их флуореценции в закрытой пробирке. Для дальнейших экспериментов была собрана экспериментальная установка, которая позволила проводить измерение флуресцении в течении каждого термического цикла и получать кинетические кривые флуоресцении [12]. Такие данные позволяют не только подтвердить наличие интересующей ДНК в образце, но и оценить начальное количество ДНК.
"Кинетическая" ПЦР стала последним недостающим ключом для входа ПЦР в диагностическую область. Возможность проведения ПЦР и детектирования продуктов в закрытой пробирке значительно упростило анализ и увеличило надежность получаемых результатов. Теперь можно проводить масштабные и потоковые исследования не боясь контаминации лаборатории и реактивов и не беспокоясь за правильность получаемых результатов.
Мультиплексная ПЦР
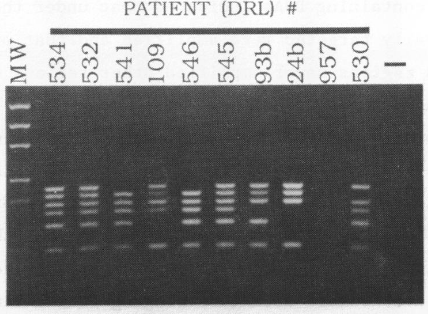
Паралельно с развитием кинетической пцр шло развитие мультиплексной ПЦР - варианта ПЦР, при котором проводилась амплификация нескольких участков ДНК в одном растворе. В 1988 г. Томас Кески опубликовал работу, в которой проводился скрининг геномной ДНК на наличие делеций [13]. Идентификация ампликонов проводилось электрофоретически, поэтому длины амплифицируемых участков выбирались так, чтобы быть различимы в геле.
Практическая значимость мультиплексной ПЦР сразу была оценена исследователями и разработчиками:
- возможность проводить скрининг образоцов, количество которых ограничено;
- возможность выявить ложноотрицательные результаты за счет амплификации контрольного участка ДНК.
В результате работ по совершенствованию мультиплексной ПЦР появились флуоресцентные зонды, которые позволили детектировать амплификацию определенного участка ДНК. Использование нескольких флуоресцентных зондов с разными спектральными характристиками и разными нуклеотидными последовательностями позволяют раздельно детектировать несколько ампликонов в одном растворе в режиме реального времени.
Хотя Муллис и признается автором ПЦР, над созданием той ПЦР, которую мы сейчас знаем, работало много людей, которые решили целый ряд принципиальных практических ограничений. Так или иначе, с момента открытия ПЦР прошла долгий путь развития для того чтобы занять текущее место в генетическом анализе.
Цифровая ПЦР
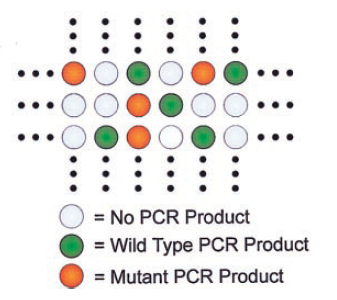
В некоторых практических задачах высокой селективности ПЦР недостаточно. Для определения единичной мутации в единичных молекулах ДНК на фоне высокого избытка ДНК дикого типа применяется техника "цифровой ПЦР", когда исходную пробу разбавляют до такой степени, что в реактор попадает лишь одна молекула дикого типа [14]. В это случае происходит несколько эффектов:
- Селективноe концентрированиe целевой молекулы. Например, раствор содержит 5 целевых молекул и 5000 мешающих. После разбавления и аликвотирования в 200 реакторов в 5 реакторах оказывается по 1 целевой молекулы. Мешающие молекулы распределяются равномерно по 25 молекул. В результате в начале доля целевых молекул составляет 0,1%, а после разбавления - 4%, произошло концентрирование в 40 раз.
- Возможен количественный анализ единичных молекул ДНК. При классическом количественном ПЦР растворы с низким содержанием ДНК характеризуются большим пороговым циклом, соответственно, неидеальность амплификации (эффективность ПЦР не равна 2) и, главное, ее непостоянство оказывают значительное влияние на точность количественной оценки. Дополнительно, логарифмический характер градуировочной зависимости значительно увеличивает погрешность анализа при прямом представлении результатов. В случае же цифровой ПЦР количество реакторов с положительным результатом соответствует количеству молекул ДНК [15].
ПЦР с обратной транскрипцией
Область применения ПЦР можно расширить и на молекулы РНК за счет реации обратной транскрипции, в ходе которой обратная транскриптаза по матрице РНК строит комплементарную ДНК. Далее эта ДНК используется как матрица в ПЦР [16]. ПЦР с обратной транскрипцией можно проводить последовательно в две стадии в двух растворах, и одновременно в одном растворе [17]. В качестве затравок для обратной транскриптазы используются могут использоваться как специфичные праймеры (аналогично ПЦР), так и неспецифичные ("вырожденные", или рандомные, праймеры или олиго(dT)-праймеры). Практическое применения ОТ-ПЦР нашла в диагностике РНК содержащих вирусов и изучении экспрессии генов.
Литература
- R .Lehman, M. Bessman,E. Simms, A. Kornberg / Enzymatic Synthesis of Deoxyribonucleic Acid. I. Preparation of substrates and partial purification of an enzyme from escherichia coli // Published in Harvey lectures, 1957
- K. Kleppe, E. Ohtsuka, R. Kleppe, I. Molineux, H. Khorana / Repair replication of short synthetic DNA's as catalyzed by DNA polymerases // J. Molecular biology. Biol., 1971, 56, 341-361
- A. Chien, D. Edgar, J Trela / Deoxyribonucleic asid polymerase from the extreme thermophile Thermus aquatics // J. Bacteriology, 1976, 127(3) 1550-1957
- J. Bartlett, D. Stirling / PCR Protocols, second edition // Humana Press Inc., 2003
- P. Rabinow / Making PCR: A Story of Biotechnology // University of Chicago Press, 1997
- K. Olson, T. Gabriel, J. Michalewsky, C. Harvey / Enzymatic multiplication of a chemically synthesized DNA fragment // Nucleic Acids Research, 1975, 2(1), 43-60
- A. Panet, H. Khorana / The linkage of deoxyribopolynucleotide templates to cellulose and its use in their replication // The Journal of Biological Chemistry, 1974, 249, 5213-5221
- K. Mullis, F. FerréRichard, A. Gibbs / The Polymerase Chain Reaction // Birkhäuser Boston, 1994
- K. Mullis, F. Faloona, S. Scharf, R. Saiki, G. Horn, H. Erlich / Specific Enzymatic Amplification of DNA In Vitro: The Polymerase Chain Reaction // Cold Spring Harbor Symposia on Quantitative Biology, Cold Spring Harbor Laboratory, 1986
- R. Saiki, D. Gelfand, S. Stoffel, S. Scharf, R. Higuchi, G. Horn, K. Mullis, H. Erlich / Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. // Science, 1988, 239, 487-491
- T.R. Gingeras, R. Higuchi, L.J. Kricka, Y.M. Lo, C.T. Wittwer / Fifty years of molecular (DNA/RNA) diagnostics // Clinical Chemistry, 2005, 51(3), 661-671
- R. Higuchi, C. Fockler, G. Dollinger, R. Watson / Kinetic PCR analysis: real-time monitoring of DNA amplification reactions // Biotechnology, 1993, 11(9), 1026-30
- J. Chamberlain, R. Gibbs, J. Rainer, P. Nga Nguyen, T. Caskey / Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification // Nucleic Acids Research, 1988, 16(23), 11141–11156
- B. Vogelstein, K. Kinzler / Digital PCR // Genetics, 1999, 96, 9236–9241
- K. Heyries, C. Tropini, M. VanInsberghe, C Doolin,O. Petriv, A. Singhal, K. Leung, C. Hughesman, C. Hansen / Megapixel digital PCR // Nature Methods, 2011, 8(8), 649-651
- D. Rappolee, D Mark, M. Banda, Z Werb / Wound macrophages express TGF-alpha and other growth factors in vivo: analysis by mRNA phenotyping // Science, 1988, 241, 708-712
- S. Lee, P. Chang, J. Shien, M. Cheng, H. Shieh / Identification and subtyping of avian influenza viruses by reverse transcription-PCR // Journal of Virological Methods, 2001, 97, 13–22
